Cook Abstand
Eine Möglichkeit Ausreißer zu finden ist der Cook Abstand (eng.: Cook’s distance). Die Idee ist es zu messen welchen Einfluss ein Wert auf das Modell hat. Dazu schaut mich sich das Modell einmal mit und einmal ohne den Wert an und vergleicht diese Ergebnisse.
Schauen wir uns den Cook Abstand einmal für ein (einfaches) lineares Regressionmodell konkret an:
Vorbereitungen
Wir wollen mit mosaic arbeiten, also laden wir das Paket als erstes:
library(mosaic)Falls die tipping-Daten noch nicht im Verzeichnis liegen, laden wir diese aus dem Internet nach:
if (!file.exists("tips.csv")) {
download.file("https://goo.gl/whKjnl", destfile = "tips.csv")
}Nun laden wir die tipping-Daten in den Speicher in den Datenrahmen tips:
tips <- read.csv2("tips.csv")Wir brauchen für unser Modell nur den Rechnungsbetrag total_bill und den
Trinkgeldbetrag tip für unser Modell:
tips %>% select(c("total_bill", "tip")) -> tipsUnser Modell:
gf_point(tip ~ total_bill, data = tips)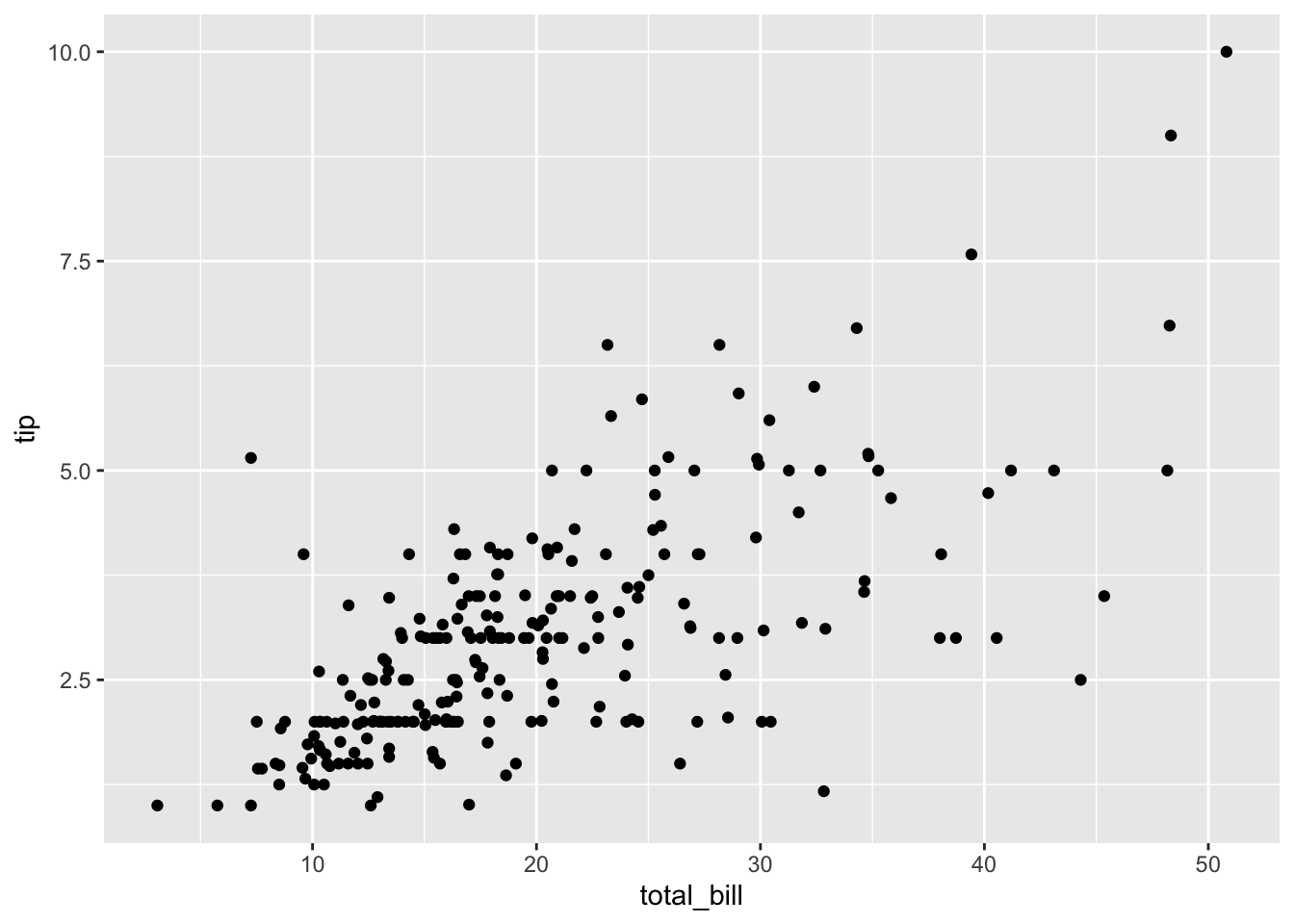
erg_lm <- lm(tip ~ total_bill, data = tips)
summary(erg_lm)##
## Call:
## lm(formula = tip ~ total_bill, data = tips)
##
## Residuals:
## Min 1Q Median 3Q Max
## -3.1982 -0.5652 -0.0974 0.4863 3.7434
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.920270 0.159735 5.761 2.53e-08 ***
## total_bill 0.105025 0.007365 14.260 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.022 on 242 degrees of freedom
## Multiple R-squared: 0.4566, Adjusted R-squared: 0.4544
## F-statistic: 203.4 on 1 and 242 DF, p-value: < 2.2e-16Betrachten wir nun die Regressionsgerade in unseren Daten:
gf_point(tip ~ total_bill, data = tips) +
geom_line(aes(x = total_bill,
y = erg_lm$fitted.values,
color = "Regressionsgerade"))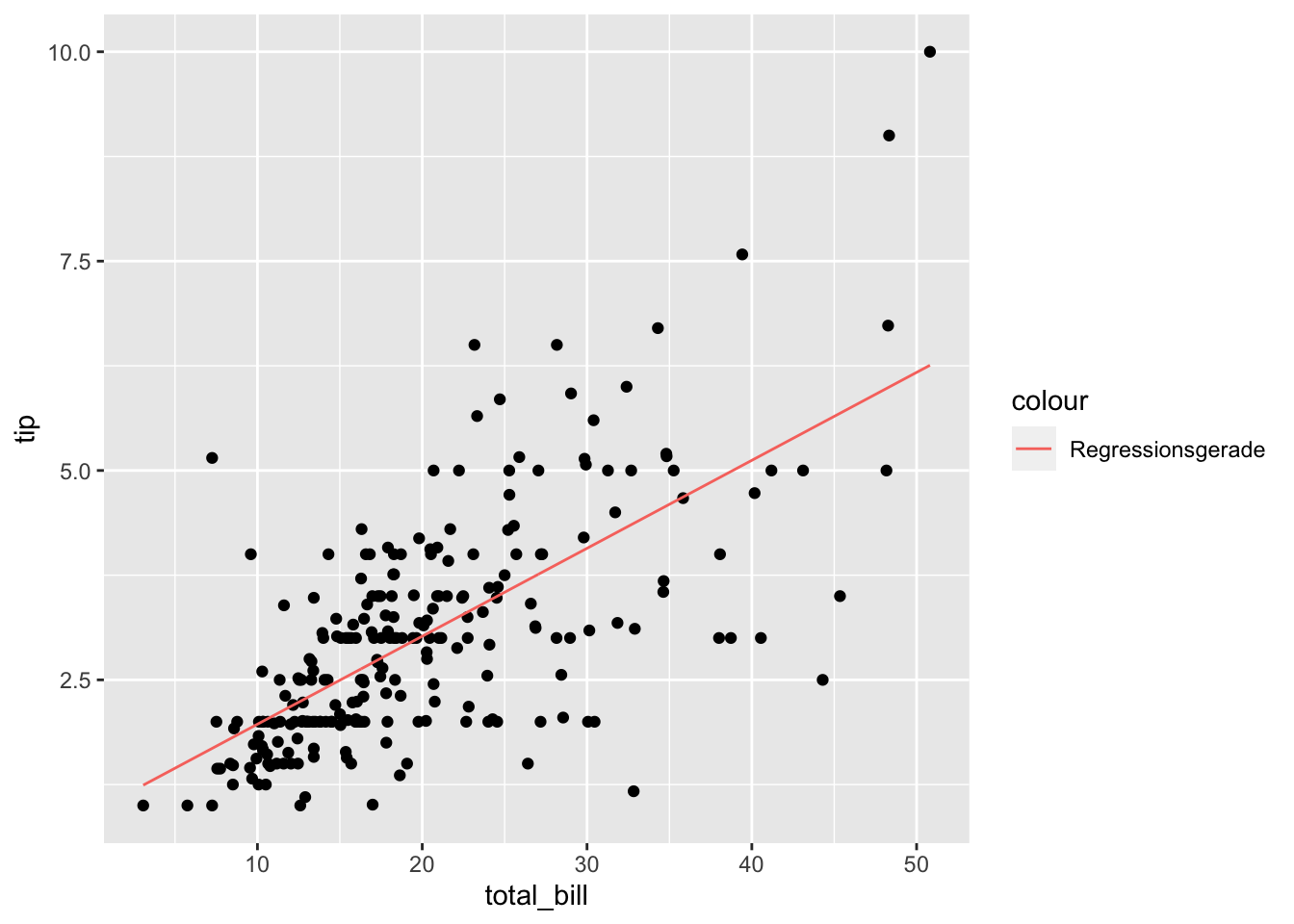
Für lineare Regressionsmodell können einflussreiche Ausreißer sehr hinderlich sein. Was also ändert sich nun, wenn wir einen Wert, z.B. einen Ausreißer, nicht betrachten?
Als Beispiel wählen wir die folgende Beobachtung aus:
tips %>% slice(173) -> tips_removed
tips_removed## total_bill tip
## 1 7.25 5.15tips %>% slice(-173) -> tips_red
erg_lm_red <- lm(tip ~ total_bill, data = tips_red)
summary(erg_lm_red)##
## Call:
## lm(formula = tip ~ total_bill, data = tips_red)
##
## Residuals:
## Min 1Q Median 3Q Max
## -3.2136 -0.5351 -0.0818 0.4951 3.6869
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.86065 0.15709 5.479 1.08e-07 ***
## total_bill 0.10731 0.00723 14.843 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.9992 on 241 degrees of freedom
## Multiple R-squared: 0.4776, Adjusted R-squared: 0.4754
## F-statistic: 220.3 on 1 and 241 DF, p-value: < 2.2e-16gf_point(tip ~ total_bill, data = tips_red) +
geom_line(aes(x = total_bill,
y = erg_lm_red$fitted.values,
color = "Regressionsgerade")) +
geom_point(aes(x = tips_removed$total_bill,
y = tips_removed$tip,
colour = "Removed Data",
show.legend = FALSE))## Warning: Ignoring unknown aesthetics: show.legend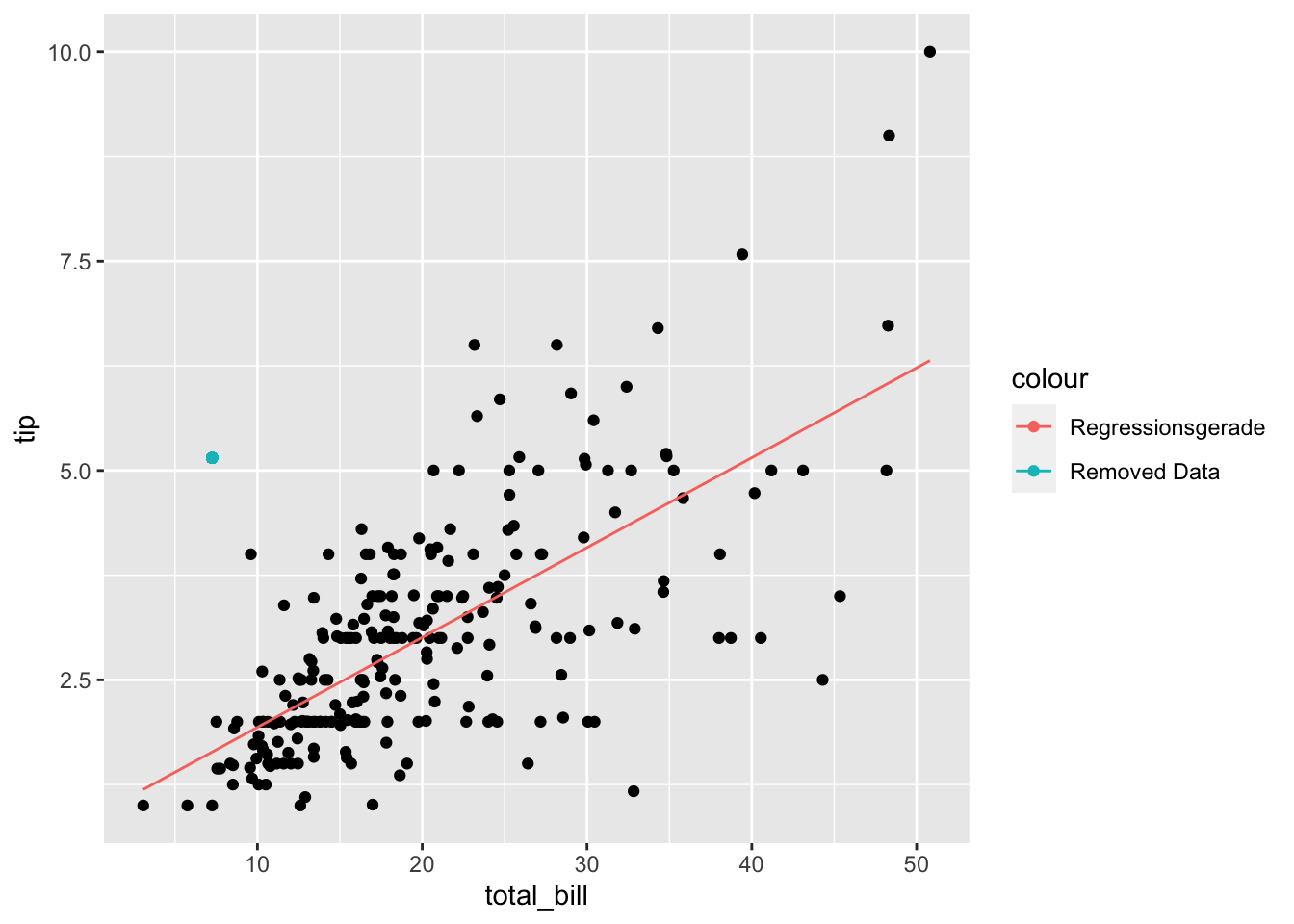
Um zu messen, was diese Änderung bewirkt hat, schaut sich der Cook Abstand zunächst die Summe der quadrierten Differenzen der vorhergesagten Werte in beiden Modellen an:
new_data <- data_frame(total_bill = tips$total_bill)## Warning: `data_frame()` is deprecated as of tibble 1.1.0.
## Please use `tibble()` instead.
## This warning is displayed once every 8 hours.
## Call `lifecycle::last_warnings()` to see where this warning was generated.prognose_lm <- predict(erg_lm, newdata = new_data)
prognose_lm_red <- predict(erg_lm_red, newdata = new_data)\[d_j = \sum_{i=1}^n \left(\hat{y}_i - \hat{y}_{i(j)}\right)^2\] Dabei ist \(\hat{y}_i\) die Prognose des Wertes \(y_i\) auf Basis von \(x_i\) mit dem Originalmodell und \(\hat{y}_{i(j)}\) die Prognose wenn man die \(j\)-te Beobachtung aus dem Modell gestrichen hat.
d_j <- sum((prognose_lm - prognose_lm_red)^2)
d_j## [1] 0.1511406Der Cook Abstand \(D_j\) wird nun noch normiert durch \[var_{cook} = p \cdot s_{\epsilon_i^2}^2\] Dabei ist \(s_{\epsilon_i^2}^2\) der erwartungstreue Schätzer der Varianz der Residuen und \(p\) die Anzahl aller erklärenden Variablen (hier \(1\)) \(+ 1\).
Es ist also:
\[D_j = \frac{d_j}{var_{cook}} = \frac{\sum\limits_{i=1}^n \left(\hat{y}_i - \hat{y}_{i(j)}\right)^2}{p \cdot s_{\epsilon_i^2}^2}\]
# Summary des Modells
selm <- summary(erg_lm)
# Wir finden p als rank im Modell
p <- erg_lm$rank
# Wir finden den erwatungtreuen Schätzer im Summary des Modells
s_quad_eps_quad <- (selm$sigma)^2
var_cook = p * s_quad_eps_quad
D_j = d_j / var_cook
D_j## [1] 0.07234504Wir können den Wert aber auch viel einfacher direkt berechnen lassen und dass
für alle \(j\) mit Hilfe von cooks.distance(..):
cooks.distance(erg_lm)[173]## 173
## 0.07234504Wann aber ist nun ein Wert ein einflussreicher Ausreißer?
Cook selber gibt dafür die Bedingung \(D_j > 1\) an. Andere Autor*innen schreiben \(D_j > 4/n\), wobei \(n\) die Anzahl der Beobachtung ist.
In unserem Beispiel liefert die Variante \(D_j > 1\)
cooks <- cooks.distance(erg_lm)
names(cooks) <- NULL
n <- nrow(tips)
any(cooks > 1)## [1] FALSEkeinen Ausreißer.
Wenn wir jedoch mit \(D_j > 4/n\) suchen .
any(cooks > 4/n)## [1] TRUEdann gibt es Ausreißer. Die Indices dieser finden wir mit:
which(cooks > 4/n)## [1] 24 48 57 103 142 157 171 173 179 183 184 185 188 208 211 213 215 238Bereinigen wir nun unsere Daten um genau diese Werte:
remove <- which(cooks > 4/n)
tips %>% slice(-remove) -> tips_no_outliers
erg_lm_no_outliers <- lm(tip ~ total_bill, data = tips_no_outliers)Und schauen uns das Ergebnis an:
summary(erg_lm_no_outliers)##
## Call:
## lm(formula = tip ~ total_bill, data = tips_no_outliers)
##
## Residuals:
## Min 1Q Median 3Q Max
## -2.22592 -0.48166 -0.06794 0.46992 2.31414
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.773324 0.139435 5.546 8.2e-08 ***
## total_bill 0.111799 0.006958 16.069 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.7778 on 224 degrees of freedom
## Multiple R-squared: 0.5355, Adjusted R-squared: 0.5334
## F-statistic: 258.2 on 1 and 224 DF, p-value: < 2.2e-16gf_point(tip ~ total_bill, data = erg_lm_no_outliers) +
geom_line(aes(x = total_bill,
y = erg_lm_no_outliers$fitted.values,
color = "Regressionsgerade"))
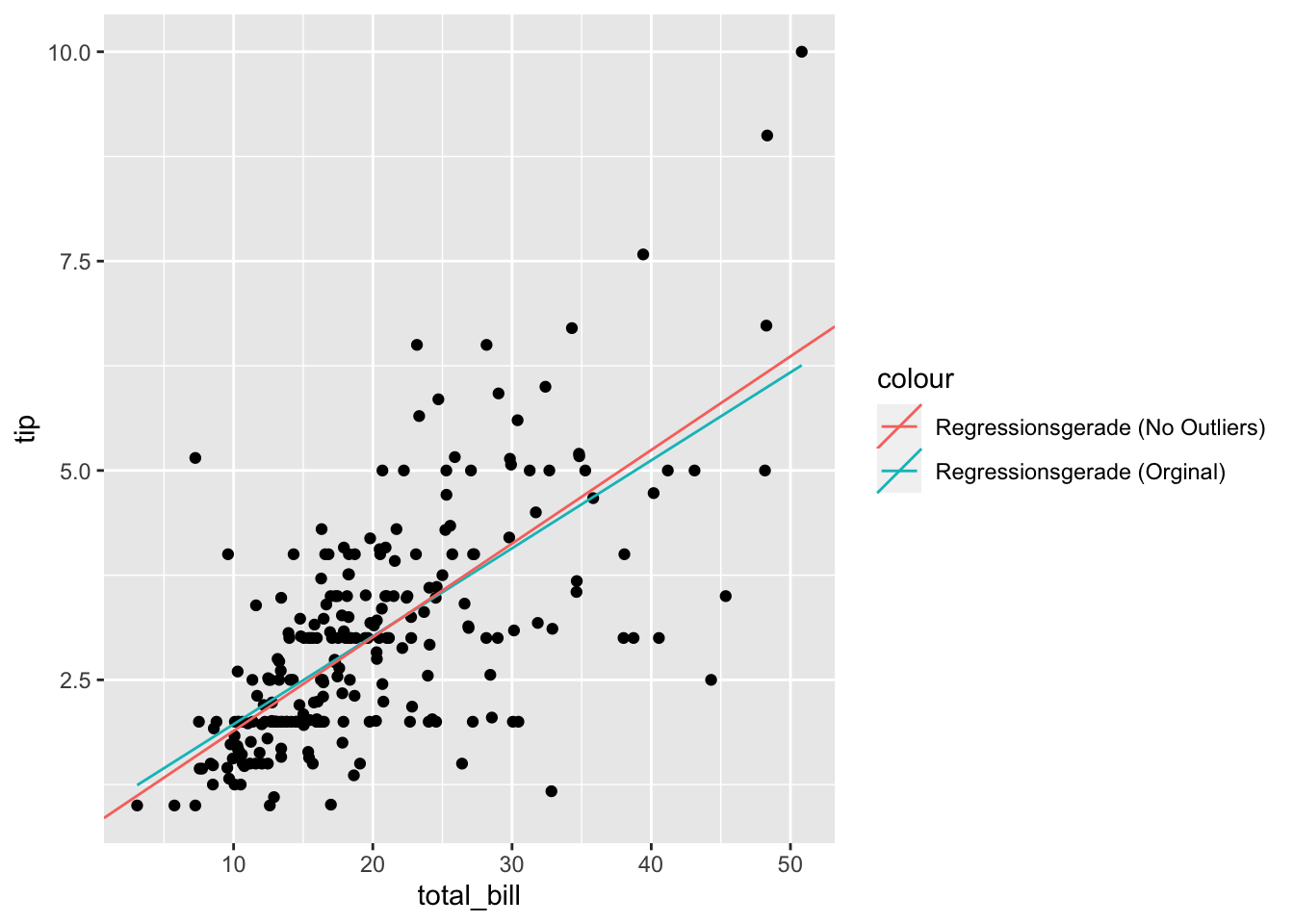
Im direkten Vergleich:
gf_point(tip ~ total_bill, data = erg_lm) +
geom_line(aes(x = total_bill,
y = erg_lm$fitted.values,
colour = "Regressionsgerade (Orginal)")) +
geom_abline(aes( colour = "Regressionsgerade (No Outliers)",
intercept = erg_lm_no_outliers$coefficients[1],
slope = erg_lm_no_outliers$coefficients[2])
)